- ОРДИ
- Каналы
- Межрегиональная ассоциация по клинической микробиологии и антимикробной химиотерапии (МАКМАХ)
- AMRmap: Интернет-платформа мониторинга антибиотикорезистентности
AMRmap дает возможность получения актуальной информации об антибиотикорезистентности, что может помочь
в рациональном выборе антимикробных химиопрепаратов при инфекциях различной локализации.
Авторы: Кузьменков А.Ю., Трушин И.В., Авраменко А.А., Эйдельштейн М.В., Дехнич А.В., Козлов Р.С.
НИИ антимикробной химиотерапии ФГБОУ ВО СГМУ Минздрава России, Смоленск, Россия
Клиническая Микробиология и Антимикробная Химиотерапия. 2017; 19(2):84-90
Изменение уровня устойчивости микроорганизмов к антимикробным препаратам (АМП) в пространстве и времени предопределяет важность мониторинга антибиотикорезистентности, что в свою очередь имеет ключевое значение для оптимизации антибиотикотерапии и мер инфекционного контроля. Однако, в большинстве случаев результаты мониторинга антибиотикорезистентности представляются в виде публикаций в научных журналах с отставанием до двух лет, что закономерно снижает ценность полученных результатов в решении задач оперативного реагирования, а разрозненность публикаций затрудняет оценку трендов. Кроме того, сам по себе формат представления данных на бумажных носителях зачастую ограничивает авторов в полноценности представления данных. Решение данных проблем стало возможным благодаря развитию IT-технологий и появлению так называемых «интерактивных» систем мониторинга антибиотикорезистентности. В мире существуют такие системы мониторинга антибиотикорезистентности как EARS-Net, CDDEP ResistanceMap, SGSS, NNIS system, ATLAS, SMART, в которых заложена возможность интерактивного анализа и/или представления данных [1–6]. Тем не менее, указанные системы имеют ряд серьезных ограничений, связанных с фильтрацией, визуализацией и анализом данных, подробный разбор которых заслуживает отдельного обсуждения. Целью же настоящего обзора является описание основных возможностей и принципов работы с интернет-ресурсом AMRmap (map.antibiotic.ru) – системы мониторинга антибиотикорезистентности в Российской Федерации, учитывающей опыт использования и ограничения уже существующих аналогичных систем.
AMRmap является веб-приложением со свободным доступом, расположенным по адресу http://map.antibiotic.ru.
Веб-приложение представляет данные, полученные при проведении многоцентровых исследований антибиотикорезистентности НИИ антимикробной химиотерапии ФГБОУ ВО СГМУ Минздрава России совместно сМежрегиональной ассоциацией по клинической микробиологии и антимикробной химиотерапии (МАКМАХ). Постоянно пополняемая и обновляемая база данных в настоящий момент содержит информацию о чувствительности к широкому спектру антибиотиков более 40 тыс. клинических изолятов бактерий из 72 городов Российской Федерации за период 1997 по 2016 гг.
· Перейдя по ссылке http://map.antibiotic.ru, пользователь попадает на главную страницу приложения (рис. 1). В верхней части экрана расположены навигационные вкладки, переключаясь между которыми, пользователь может выбрать интересующий раздел приложения (рис. 1). На странице «Главная» пользователю доступна возможность активации мобильной версии приложения, которая оптимизирует работу на устройствах Android и iOS. Приложение предоставляет два уровня прав доступа: общий (получают все пользователи по умолчанию) и экспертный. Пользователям с общими правами доступен анализ данных до уровня отдельных городов, для пользователей с экспертным доступом – до уровня отдельных ЛПУ.
· Для начала работы с приложением необходимо перейти на страницу «Анализ данных» путем нажатия на соответствующую надпись в верхней части экрана, либо путем нажатия на кнопку «Начать работу». После этого откроется страница «Анализ данных», в которой условно можно выделить две составляющих: поле параметров и поле инфографики (рис. 2).
· С помощью поля параметров приложение обеспечивает возможность фильтрации данных по: источнику возникновения инфекции (внебольничная/нозокомиальная), географической локализации, типу и локализации инфекции, виду клинического материала, профилю отделения, возрасту пациента, группе/ видам микроорганизмов (для всех параметров доступен текстовый поиск и множественный выбор). Заполняя последовательно параметры от 1 до 10 (рис. 2) и нажав кнопку «Отобразить ТОП 10» пользователю станут доступны интерактивные диаграммы распределения групп и видов микроорганизмов по частотевстречаемости для выбранных параметров (рис. 3).
· Поле инфографики предназначено для анализа и отображения данных об устойчивости выбранного микроорганизма(-ов) к различным антимикробным препаратам (АМП). Поле инфографики включает в себя три аналитических модуля: «Основная информация», «Перекрестная устойчивость», «Генетические детерминанты резистентности» (рис. 2).
· После перехода в модуль «Основная информация» пользователю станут доступными вкладки «Антибиотики (все)» и «Выбранный антибиотик».
· При нажатии на вкладке «Антибиотики (все)» кнопки «Отобразить», происходит построение столбчатой интерактивной гистограммы с 95% ДИ, отражающей уровень устойчивости выбранного микроорганизма(-ов) ко всем доступным АМП (рис. 4).
· При переходе на вкладку «Выбранный антибиотик» пользователю станет доступен ряд подвкладок («Суммарная информация», «Гистограмма», «Гистограмма+95% ДИ», «Таблица», «Карта», «Тренды», «Регрессия», «МПК»), предназначенных для анализа пространственно-временной изменчивости уровня устойчивости к конкретному АМП (рис. 5).
· Для начала работы с данными подвкладками необходимо выбрать интересующий АМП. После выбора АМП и нажатия кнопки «Отобразить» на подвкладках станут доступными различные графики и таблицы среди них: интерактивные таблицы и диаграммы распределения изолятов по категориям чувстви- тельности (стакбары или барплоты с указанием доверительных интервалов), диаграммы распределения изолятов по уровням устойчивости к антибиотикам (значениям МПК); интерактивные карты с указанием абсолютных и относительных показателей распространенности резистентности; графики трендов изменения чувствительности/резистентности с оценкой значимости различий между отдельными периодами; графики регрессии с возможностью визуализации распределения показателей резистентности в разных городах относительно общей линии тренда и анализа «выпадающих точек» с аномально высокими или низкими показателями устойчивости.
· Аналитический модуль «Перекрестная устойчивость» предназначен для анализа показателей перекрестной и ассоциированной устойчивости к различным АМП. При переходе в данный модуль становятся доступными ряд вкладок: «Матрица», «Граф», «Тренд», «Таблица».
· На вкладке «Матрица» отображается интерактивная матрица перекрестной устойчивости, где число в каждой ячейке означает процент изолятов, нечувствительных к АМП
по столбцу, от общего количества изолятов, нечувствительных к АМП по строке (рис. 6).
· На вкладке «Граф» и «Таблица» отображается информация о перекрестной и ассоциированной устойчивости
в виде интерактивного графа и таблицы соответственно.
· На вкладке «Тренд» отображается перекрестной
устойчивости по двум выбранным в этой же вкладке
АМП (рис. 7).
· Модуль «Генетические детерминанты резистентности» предназначен для анализа распространения важнейших детерминант антибиотикорезистентности. На настоящий момент доступна информация о генах, приобретенных карбапенемаз (групп: NDM, VIM, IMP, KPC, GES-2/5, OXA-48, OXA-23, OXA24/40, OXA-58) у грамотрицательных возбудителей (Enterobacteriaceae, Pseudomonas и Acinetobacter spp.). Данныйаналитический модуль включает ряд вкладок («Карта», «Маркеры», «Антибиотики (все)», «МПК», «Тренд», Время первого обнаружения»).
· Вкладка «Карта» предназначена для анализа распространения генетических детерминант резистентности, а на вкладке «Маркеры» представлена суммарная информация по частоте и распространенности генетических детерминант резистентности.
· Фенотипические данные, представленные на вкладке «Антибиотики (все)», соответствуют данным на одноименной вкладке из аналитического модуля «Основная информация» с той лишь разницей, что проанализированы для изолятов с наличием выбранных пользователем генетических детерминант.
· На вкладке «МПК» отображается интерактивный график распределения МПК для выбранного АМП с учетом генетических детерминат устойчивости (рис. 8).
· Вкладка «Тренд» демонстрирует абсолютное количество изолятов с различными генетическими детерминантами устойчивости по годам. Вкладка «Время первого обнаружения» предназначена для анализа и визуализации пространственно-временного распространения выбранного генетического маркера устойчивости с помощью интерактивной временной шкалы, расположенной здесь же на подвкладке «Шкала» и специальной интерактивной карты, расположенной на под- вкладке «Карта» (рис. 9-10).
В будущем планируется получение данных из локальных микробиологических лабораторий с возможностью их автоматической валидации и представления онлайн в режиме реального времени, а также создание дополнительных аналитических модулей. Создание и развитие специализированных интернет-ресурсов открывает новые возможности для организации комплексного оперативного мониторинга состояния АБР в РФ. AMRmap дает возможность получения актуальной информации об антибиотикорезистентности, что может помочь в рациональном выборе антимикробных химиопрепаратов при инфекциях различной локализации.
Литература
1. MSD. SMART: Study For Monitoring Antimicrobial Resistance Trends. Available at: http://www.globalsmartsite.com. Accessed: 11.07.2016.
2. Pfizer. ATLAS: Antimicrobial Testing Leadership and Surveillance. Available at: https://atlas-surveillance.com. Accessed: 01.05.2017.
3. CDC. Antibiotic Resistance Patient Safety Atlas – Data on Antibiotic-Resistant Healthcare-Associated Infections. Available at: http://gis.cdc.gov/grasp/ PSA/MapView.html. Accessed: 09.12.2016.
4. CDDEP. ResistanceMap. Antibiotic Resistance. Available at: https:// resistancemap.cddep.org/AntibioticResistance.php. Accessed: 12.12.2015.
5. ECDC. European Antimicrobial Resistance Surveillance Network (EARS- Net). Available at: http://ecdc.europa.eu/en/healthtopics/antimicrobial- resistance-and-consumption/antimicrobial_resistance/EARS-Net/Pages/ EARS-Net.aspx. Accessed: 11.07.2016.
6. Public Health England. Second Generation Surveillance System (SGSS). Available at: https://sgss.phe.org.uk/Security/Register. Accessed: 09.12.2016.

Рисунок 1. Стартовая страница AMRmap (map.antibiotic.ru)
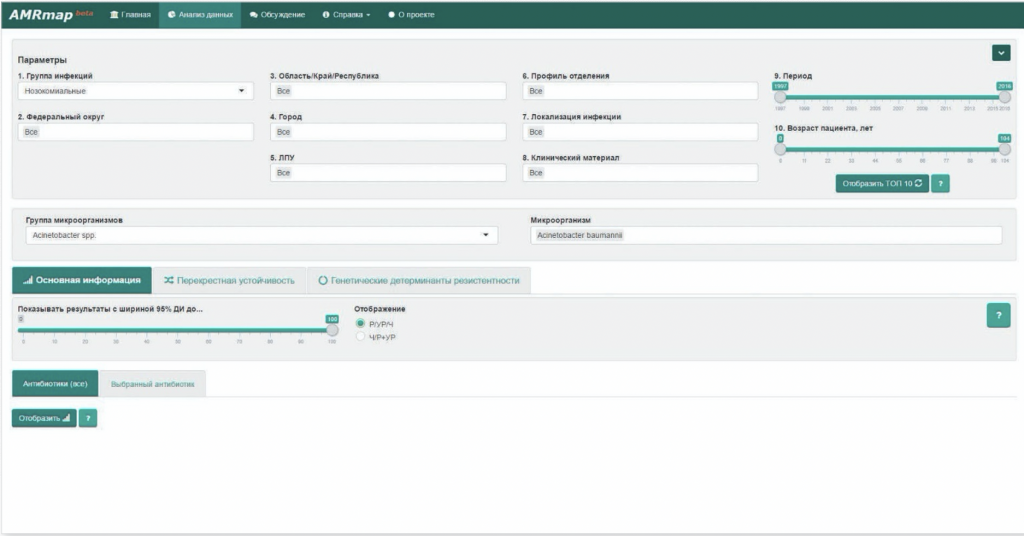
Рисунок 2. Страница «Анализ данных» AMRmap

Рисунок 3. Гистограмма распределения групп и видов микроорганизмов по частоте встречаемости (параметры выбраны по умолчанию)

Рисунок 4. Уровень устойчивости A. baumannii ко всем доступным антимикробным препаратам в приложении AMRmap

Рисунок 5. Вкладка «Выбранный антибиотик»: устойчивость A. baumannii к имипенему

Рисунок 6. Матрица перекрестной и ассоциированной устойчивости для A. baumannii
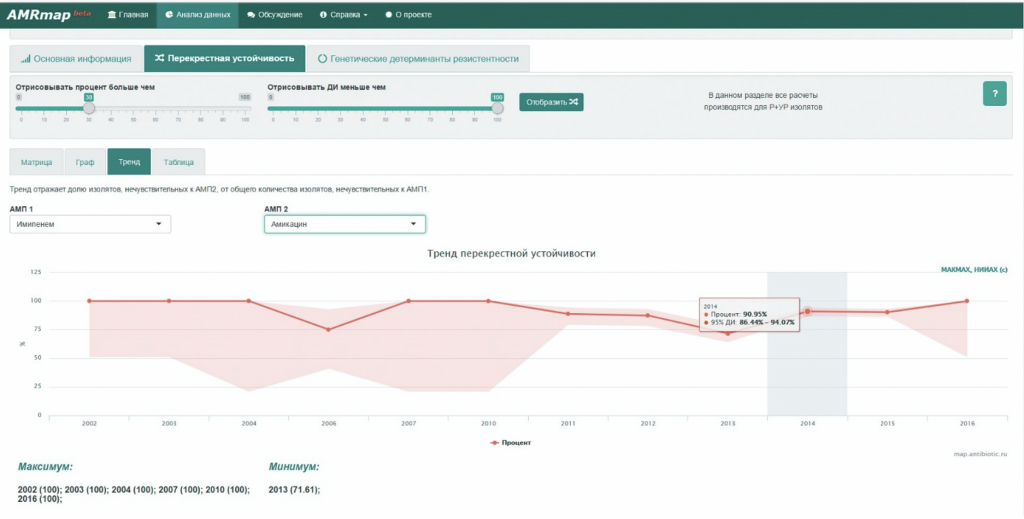
Рисунок 7. Тренд перекрестной и ассоциированной устойчивости A. baumannii к имипенему и амикацину

Рисунок 8. Распределение значений МПК имипенема для A. baumannii с учетом генетических детерминант устойчивости

Рисунок 9. Распространение OXA-23-группы карбапенемаз среди изолятов A. baumannii. Интерактивная временная шкала
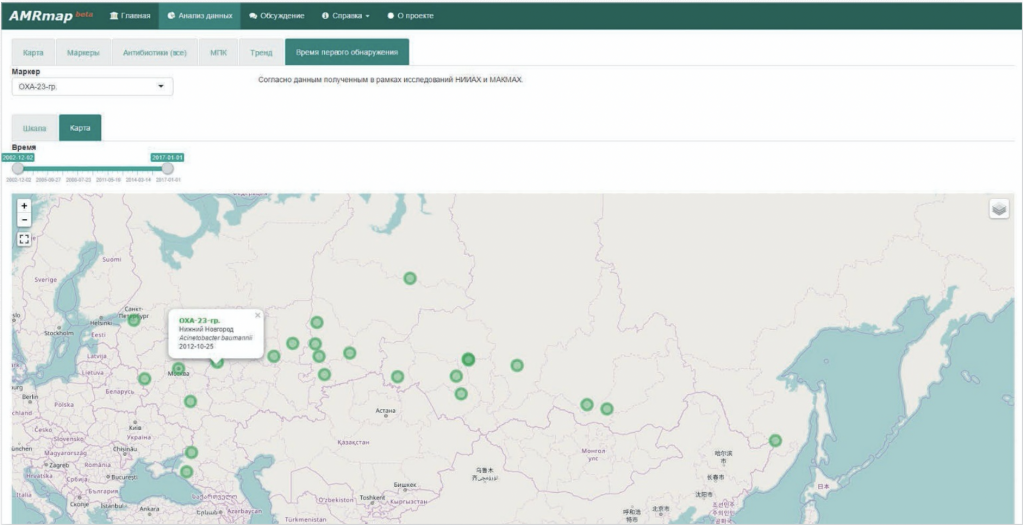
Рисунок 10. Распространение OXA-23-группы карбапенемаз среди изолятов A. baumannii – интерактивная карта
оценка


